高考生物二轮复习限时集训:14(B)基因工程(含解析)
文档属性
| 名称 | 高考生物二轮复习限时集训:14(B)基因工程(含解析) |  | |
| 格式 | docx | ||
| 文件大小 | 704.9KB | ||
| 资源类型 | 试卷 | ||
| 版本资源 | 通用版 | ||
| 科目 | 生物学 | ||
| 更新时间 | 2024-02-15 22:04:47 | ||
图片预览

文档简介
高考生物二轮复习限时集训
14(B) 基因工程
[时间:20min]
1.[2023·福建厦门模拟] 下表为四种限制酶所识别的核苷酸序列及相应的切割位置(箭头所指),下列叙述正确的是 ( )
限制 酶种类 序列及切点 限制 酶种类 序列及切点
①EcoRⅠ ↓ 5'—GAATTC—3' 3'—CTTAAG—5' ↑ ③BglⅡ ↓ 5'—AGATCT—3' 3'—TCTAGA—5' ↑
②BamHⅠ ↓ 5'—GGATCC—3' 3'—CCTAGG—5' ↑ ④NotⅠ ↓ 5'—GCGGCCGC—3' 3'—CGCCGGCG—5' ↑
A.①②③④可催化磷酸二酯键和氢键断裂,形成黏性末端
B.①切出的 DNA 片段,只有用E.coliDNA连接酶才能连接起来
C.②③切出的末端连接后形成的片段,不能再被②或③识别切割
D.构建基因表达载体时,用②③进行双酶切,可防止目的基因与载体的反向连接
2.[2023·山东济南三模] 转化是指目的基因进入受体细胞内,并且在受体细胞内维持稳定和表达的过程。下列有关说法错误的是 ( )
A.大肠杆菌转化实验中可使用Ca2+处理以提高转化效率
B.目的基因导入受体细胞前必须构建基因表达载体以便稳定存在
C.将DNA溶液滴加在授粉后的花柱切面上,可以使目的基因进入胚囊
D.农杆菌转化法中需要进行两次体外拼接和两次导入
3.[2023·安徽安庆模拟] 黏粒是质粒的衍生物,cos序列是λ噬菌体将DNA包装到噬菌体颗粒中所需的序列。如图是黏粒作用原理的示意图,其中ori是复制原点,AmpR是氨苄青霉素抗性基因。下列相关叙述正确的是 ( )
A.线状重组黏粒进入受体菌后不会发生自身环化
B.ori和AmpR序列均便于筛选出含目的基因的受体菌
C.步骤①和②应使用同一种酶,以确保目的基因与黏粒正确连接
D.含目的基因的重组黏粒与λ噬菌体包装蛋白混合后用于构建λ噬菌体
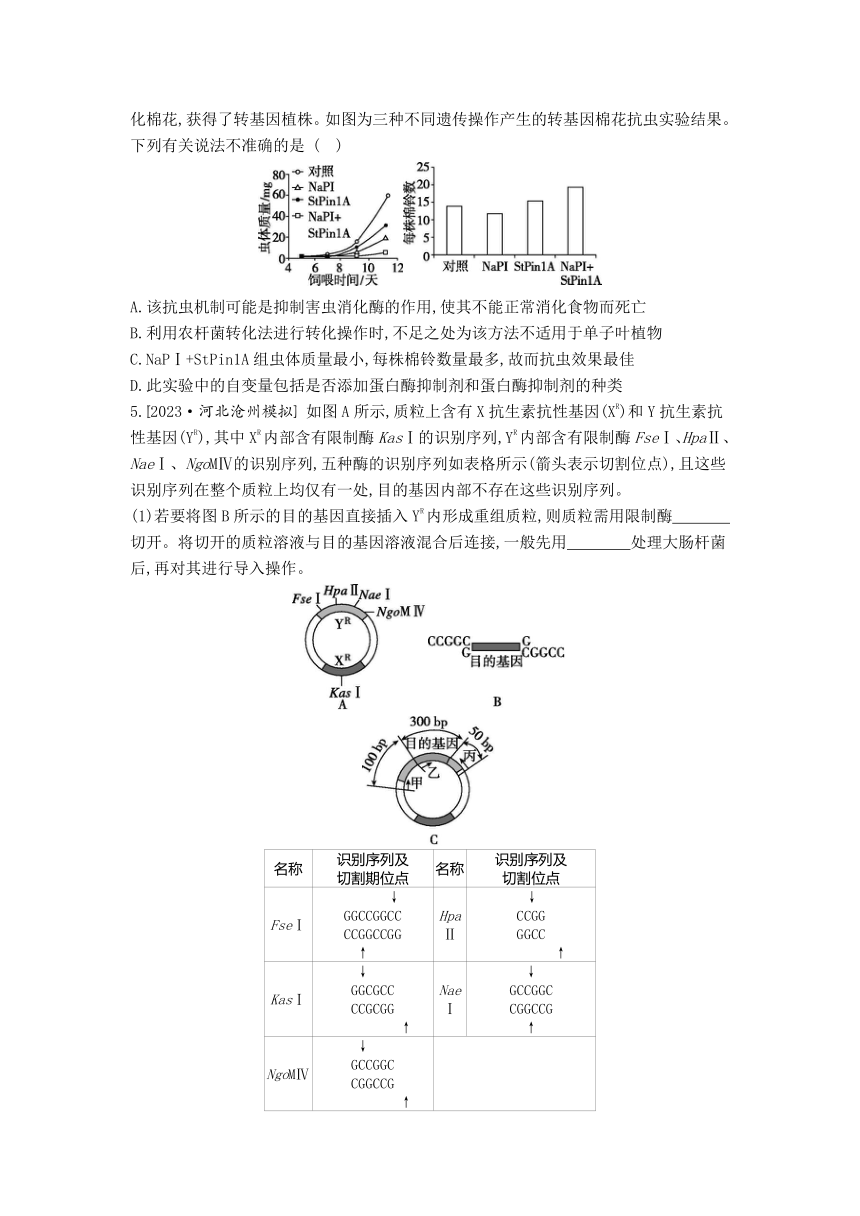
4.[2023·湖北黄冈中学二模] 蛋白酶抑制剂基因转化是作物抗虫育种的新途径。某研究团队将胰蛋白酶抑制剂(NaPⅠ)和胰凝乳蛋白酶抑制剂(StPin1A)的基因单独或共同转化棉花,获得了转基因植株。如图为三种不同遗传操作产生的转基因棉花抗虫实验结果。下列有关说法不准确的是 ( )
A.该抗虫机制可能是抑制害虫消化酶的作用,使其不能正常消化食物而死亡
B.利用农杆菌转化法进行转化操作时,不足之处为该方法不适用于单子叶植物
C.NaPⅠ+StPin1A组虫体质量最小,每株棉铃数量最多,故而抗虫效果最佳
D.此实验中的自变量包括是否添加蛋白酶抑制剂和蛋白酶抑制剂的种类
5.[2023·河北沧州模拟] 如图A所示,质粒上含有X抗生素抗性基因(XR)和Y抗生素抗性基因(YR),其中XR内部含有限制酶KasⅠ的识别序列,YR内部含有限制酶FseⅠ、HpaⅡ、NaeⅠ、NgoMⅣ的识别序列,五种酶的识别序列如表格所示(箭头表示切割位点),且这些识别序列在整个质粒上均仅有一处,目的基因内部不存在这些识别序列。
(1)若要将图B所示的目的基因直接插入YR内形成重组质粒,则质粒需用限制酶 切开。将切开的质粒溶液与目的基因溶液混合后连接,一般先用 处理大肠杆菌后,再对其进行导入操作。
名称 识别序列及 切割期位点 名称 识别序列及 切割位点
FseⅠ ↓ GGCCGGCC CCGGCCGG ↑ HpaⅡ ↓ CCGG GGCC ↑
KasⅠ ↓ GGCGCC CCGCGG ↑ NaeⅠ ↓ GCCGGC CGGCCG ↑
NgoMⅣ ↓ GCCGGC CGGCCG ↑
(2)取导入处理过的大肠杆菌接种到含有抗生素 (填“X”或“Y”)的培养基中培养,培养基 (填“需要”或“不需要”)加入琼脂。从获得的纯培养物中取部分菌体接种到含有抗生素 (填“X”或“Y”)的培养基中,若无菌落生长,可初步确定获得的培养物符合要求;若有菌落生长,原因是 。
(3)为鉴定筛选出的菌落中是否含有正确插入目的基因的重组质粒(图C所示),拟设计引物进行PCR鉴定。图C所示为甲、乙、丙3条引物在正确重组质粒中的相应位置,若选择引物 进行PCR,且扩增出了450 bp片段,则说明实验成功;若选择引物甲、乙进行PCR,且扩增出了400bp片段,原因是 。
6.[2022·山东卷] 某种类型的白血病由蛋白P引发,蛋白UBC可使P被蛋白酶识别并降解,药物A可通过影响这一过程对该病起到治疗作用。为探索药物A治疗该病的机理,需构建重组载体以获得融合蛋白FLAG-P和FLAG-P△。P△是缺失特定氨基酸序列的P,FLAG是一种短肽,连接在P或P△的氨基端,使融合蛋白能与含有FLAG抗体的介质结合,但不影响P或P△的功能。
(1)为构建重组载体,需先设计引物,通过PCR特异性扩增P基因。用于扩增P基因的引物需满足的条件是 ,为使PCR产物能被限制酶切割,需在引物上添加相应的限制酶识别序列,该限制酶识别序列应添加在引物的 (填“3'端”或“5'端”)。
(2)PCR扩增得到的P基因经酶切连接插入载体后,与编码FLAG的序列形成一个融合基因,如图甲所示,其中“ATGTGCA”为P基因编码链起始序列。将该重组载体导入细胞后,融合基因转录出的mRNA序列正确,翻译出的融合蛋白中FLAG的氨基酸序列正确,但P基因对应的氨基酸序列与P不同。据图甲分析,出现该问题的原因是 。修改扩增P基因时使用的带有EcoRⅠ识别序列的引物来解决该问题,具体修改方案是 。
(3)融合蛋白表达成功后,将FLAG-P、FLAG-P△、药物A和UBC按照图乙中的组合方式分成5组。各组样品混匀后分别流经含FLAG抗体的介质,分离出与介质结合的物质并用UBC抗体检测,检测结果如图丙所示。已知FLAG-P和FLAG-P△不能降解UBC,由①②③组结果的差异推测,药物A的作用是 ;由②④组或③⑤组的差异推测,P△中缺失的特定序列的作用是 。
(4)根据以上结果推测,药物A治疗该病的机理是 。
限时集训(十四)B
1.C [解析] ①②③④均为限制酶,可催化磷酸二酯键的断裂,不能催化氢键的断裂,且均形成黏性末端,A错误;①切出的 DNA 片段带有黏性末端,用E.coli DNA连接酶和T4DNA连接酶均能连接起来,B错误;②③切出的黏性末端相同,用DNA连接酶连接后形成的片段的碱基序列为—GGATCT—,因而不能再被②或③识别切割,C正确;构建基因表达载体时,用②③进行双酶切,不能防止目的基因与载体的反向连接,因为这两种限制酶切出的黏性末端是相同的,D错误。
2.D [解析] 可以用Ca2+处理大肠杆菌细胞,使其易于吸收周围环境中的DNA分子(使大肠杆菌处于感受态),有利于重组的基因表达载体导入其中,提高转化效率,A正确;将目的基因导入受体细胞前必须构建基因表达载体,这是基因工程的核心步骤,B正确;花粉管通道法:可以用微量注射器将含目的基因的DNA溶液直接注入子房中,也可以在植物受粉后的一定时间内,剪去柱头,将DNA溶液滴加在花柱切面上,使目的基因借助花粉管通道进入胚囊,C正确;农杆菌转化法总体上有两次拼接过程:发生在Ti质粒与目的基因之间(体外拼接)、携带目的基因的T-DNA与植物细胞的染色体DNA之间(体内拼接),两次导入过程:基因表达载体导入农杆菌、农杆菌T-DNA导入植物细胞,D错误。
3.D [解析] 若线状重组黏粒两端的黏性末端能碱基互补配对,则线状重组黏粒进入受体菌后可能会发生自身环化,A错误;ori是复制原点,不能进行筛选,AmpR序列便于筛选出含目的基因的受体菌,B错误;步骤①表示通过限制酶切割获得具有黏性末端的黏粒和目的基因,一般应使用两种限制酶,防止目的基因与黏粒反向连接,步骤②表示将黏粒和目的基因连接,应使用DNA连接酶,C错误;含目的基因的重组黏粒与λ噬菌体包装蛋白混合后用于构建λ噬菌体,然后用于侵染细菌,将目的基因导入受体菌,D正确。
4.D [解析] 胰蛋白酶和胰凝乳蛋白酶为两种消化酶,蛋白酶抑制剂的抗虫机制是阻断或减弱消化酶作用,导致害虫无法对外来蛋白进行消化作用而发育异常或死亡,A正确;农杆菌能侵染双子叶植物和裸子植物,而对大多数单子叶植物没有侵染能力,因此,利用农杆菌转化法进行转化操作时,不足之处为该方法不适用于单子叶植物,B正确;结合实验结果可看出,NaPⅠ+StPin1A组虫体质量最小,每株棉铃数量最多,故而抗虫效果最佳,C正确;由数据处理图示可以看出,此实验中的自变量有转基因植株的种类和饲喂时间,因变量是虫体质量和每株棉铃数,D错误。
5.(1)NgoMⅣ Ca2+
(2)X 需要 Y 质粒重新环化或质粒间发生自身环化,使得大肠杆菌同时具备基因XR和YR
(3)甲和丙 目的基因反向连接
[解析] (1)由目的基因形成的黏性末端可知,只有NgoM Ⅳ酶切割后的黏性末端,可以与目的基因形成的黏性末端互补配对,因此若要将图B所示的目的基因直接插入YR内形成重组质粒,则质粒需用限制酶NgoM Ⅳ切开,将切开的质粒溶液与目的基因溶液混合后连接,一般先用Ca2+处理大肠杆菌后,使其处于感受态,再对其进行导入操作。(2)质粒中的X抗生素抗性基因XR未被破坏,Y抗生素抗性基因YR由于插入目的基因而被破坏,因此取导入处理过的大肠杆菌接种到含有抗生素X的培养基中培养,为获得纯种,应在固体培养基上培养,培养基需要加入琼脂。Y抗生素抗性基因(YR)由于插入目的基因而被破坏,因此取部分菌体接种到含有抗生素Y的培养基中,若无菌落生长,可初步确定获得的培养物符合要求;若有菌落生长,说明质粒重新环化或质粒间发生自身环化,使得大肠杆菌同时具备基因XR和YR。(3)由图C可知,若要扩增出450 bp片段,且含有目的基因,只能选择引物甲和丙进行PCR。由“扩增出了400 bp片段”,即300 bp和100 bp两部分,推测可能是目的基因发生了反向连接。
6.(1)能与P基因母链的一段碱基序列互补配对、短单链核酸 5'端
(2)P基因编码链的第一个碱基与EcoR Ⅰ识别序列的最后两个碱基编码一个氨基酸,导致mRNA的密码子被错位读取 在引物中的EcoRⅠ识别序列3'端添加1个碱基
(3)增强FLAG-P与UBC的结合 参与P与UBC的结合
(4)药物A通过增强P与UBC结合促进P降解
[解析] (1)引物是一小段能与模板链的一段碱基序列互补配对的短核苷酸单链,设计扩增P基因的引物,需要两种引物分别与两条模板链3'端的碱基序列互补。DNA聚合酶延伸子链时,是将脱氧核苷酸加到引物的3'端,为了不破坏目的基因,该限制酶识别序列应添加在引物的5'端。(2)融合基因转录出的mRNA序列正确,翻译出的融合蛋白中FLAG的氨基酸序列正确,但P基因对应的氨基酸序列与P不同,说明P基因翻译时出错。由图可看出,在转录出的mRNA中,限制酶EcoRⅠ识别序列对应的最后两个碱基与P基因对应的第一个碱基构成一个密码子,导致读码框改变,翻译出的氨基酸序列改变,可通过在EcoRⅠ识别序列3'端增加1个碱基使其碱基数目加上FLAG的碱基数目为3的倍数,这样能保证P基因转录出的mRNA上的读码框不改变,能够正常翻译。(3)①组仅添加UBC,处理后,用UBC抗体检测,不出现杂交带(因为UBC不能与介质结合);②组添加UBC和FLAG-P,用UBC抗体检测,出现杂交带;③组添加UBC、药物A和FLAG-P,用UBC抗体检测,杂交带更加明显,说明药物A的作用是促进UBC与FLAG-P的结合。②④组或③⑤组的差异在于②③组使用FLAG-P,出现杂交带,④⑤组使用FLAG-P△,不出现杂交带,据此推测P△中缺失的特定序列是与UBC结合的关键序列。(4)根据(3)的分析推测,药物A促进UBC与FLAG-P的结合,从而促进蛋白P被蛋白酶识别并降解,达到治疗的目的。
14(B) 基因工程
[时间:20min]
1.[2023·福建厦门模拟] 下表为四种限制酶所识别的核苷酸序列及相应的切割位置(箭头所指),下列叙述正确的是 ( )
限制 酶种类 序列及切点 限制 酶种类 序列及切点
①EcoRⅠ ↓ 5'—GAATTC—3' 3'—CTTAAG—5' ↑ ③BglⅡ ↓ 5'—AGATCT—3' 3'—TCTAGA—5' ↑
②BamHⅠ ↓ 5'—GGATCC—3' 3'—CCTAGG—5' ↑ ④NotⅠ ↓ 5'—GCGGCCGC—3' 3'—CGCCGGCG—5' ↑
A.①②③④可催化磷酸二酯键和氢键断裂,形成黏性末端
B.①切出的 DNA 片段,只有用E.coliDNA连接酶才能连接起来
C.②③切出的末端连接后形成的片段,不能再被②或③识别切割
D.构建基因表达载体时,用②③进行双酶切,可防止目的基因与载体的反向连接
2.[2023·山东济南三模] 转化是指目的基因进入受体细胞内,并且在受体细胞内维持稳定和表达的过程。下列有关说法错误的是 ( )
A.大肠杆菌转化实验中可使用Ca2+处理以提高转化效率
B.目的基因导入受体细胞前必须构建基因表达载体以便稳定存在
C.将DNA溶液滴加在授粉后的花柱切面上,可以使目的基因进入胚囊
D.农杆菌转化法中需要进行两次体外拼接和两次导入
3.[2023·安徽安庆模拟] 黏粒是质粒的衍生物,cos序列是λ噬菌体将DNA包装到噬菌体颗粒中所需的序列。如图是黏粒作用原理的示意图,其中ori是复制原点,AmpR是氨苄青霉素抗性基因。下列相关叙述正确的是 ( )
A.线状重组黏粒进入受体菌后不会发生自身环化
B.ori和AmpR序列均便于筛选出含目的基因的受体菌
C.步骤①和②应使用同一种酶,以确保目的基因与黏粒正确连接
D.含目的基因的重组黏粒与λ噬菌体包装蛋白混合后用于构建λ噬菌体
4.[2023·湖北黄冈中学二模] 蛋白酶抑制剂基因转化是作物抗虫育种的新途径。某研究团队将胰蛋白酶抑制剂(NaPⅠ)和胰凝乳蛋白酶抑制剂(StPin1A)的基因单独或共同转化棉花,获得了转基因植株。如图为三种不同遗传操作产生的转基因棉花抗虫实验结果。下列有关说法不准确的是 ( )
A.该抗虫机制可能是抑制害虫消化酶的作用,使其不能正常消化食物而死亡
B.利用农杆菌转化法进行转化操作时,不足之处为该方法不适用于单子叶植物
C.NaPⅠ+StPin1A组虫体质量最小,每株棉铃数量最多,故而抗虫效果最佳
D.此实验中的自变量包括是否添加蛋白酶抑制剂和蛋白酶抑制剂的种类
5.[2023·河北沧州模拟] 如图A所示,质粒上含有X抗生素抗性基因(XR)和Y抗生素抗性基因(YR),其中XR内部含有限制酶KasⅠ的识别序列,YR内部含有限制酶FseⅠ、HpaⅡ、NaeⅠ、NgoMⅣ的识别序列,五种酶的识别序列如表格所示(箭头表示切割位点),且这些识别序列在整个质粒上均仅有一处,目的基因内部不存在这些识别序列。
(1)若要将图B所示的目的基因直接插入YR内形成重组质粒,则质粒需用限制酶 切开。将切开的质粒溶液与目的基因溶液混合后连接,一般先用 处理大肠杆菌后,再对其进行导入操作。
名称 识别序列及 切割期位点 名称 识别序列及 切割位点
FseⅠ ↓ GGCCGGCC CCGGCCGG ↑ HpaⅡ ↓ CCGG GGCC ↑
KasⅠ ↓ GGCGCC CCGCGG ↑ NaeⅠ ↓ GCCGGC CGGCCG ↑
NgoMⅣ ↓ GCCGGC CGGCCG ↑
(2)取导入处理过的大肠杆菌接种到含有抗生素 (填“X”或“Y”)的培养基中培养,培养基 (填“需要”或“不需要”)加入琼脂。从获得的纯培养物中取部分菌体接种到含有抗生素 (填“X”或“Y”)的培养基中,若无菌落生长,可初步确定获得的培养物符合要求;若有菌落生长,原因是 。
(3)为鉴定筛选出的菌落中是否含有正确插入目的基因的重组质粒(图C所示),拟设计引物进行PCR鉴定。图C所示为甲、乙、丙3条引物在正确重组质粒中的相应位置,若选择引物 进行PCR,且扩增出了450 bp片段,则说明实验成功;若选择引物甲、乙进行PCR,且扩增出了400bp片段,原因是 。
6.[2022·山东卷] 某种类型的白血病由蛋白P引发,蛋白UBC可使P被蛋白酶识别并降解,药物A可通过影响这一过程对该病起到治疗作用。为探索药物A治疗该病的机理,需构建重组载体以获得融合蛋白FLAG-P和FLAG-P△。P△是缺失特定氨基酸序列的P,FLAG是一种短肽,连接在P或P△的氨基端,使融合蛋白能与含有FLAG抗体的介质结合,但不影响P或P△的功能。
(1)为构建重组载体,需先设计引物,通过PCR特异性扩增P基因。用于扩增P基因的引物需满足的条件是 ,为使PCR产物能被限制酶切割,需在引物上添加相应的限制酶识别序列,该限制酶识别序列应添加在引物的 (填“3'端”或“5'端”)。
(2)PCR扩增得到的P基因经酶切连接插入载体后,与编码FLAG的序列形成一个融合基因,如图甲所示,其中“ATGTGCA”为P基因编码链起始序列。将该重组载体导入细胞后,融合基因转录出的mRNA序列正确,翻译出的融合蛋白中FLAG的氨基酸序列正确,但P基因对应的氨基酸序列与P不同。据图甲分析,出现该问题的原因是 。修改扩增P基因时使用的带有EcoRⅠ识别序列的引物来解决该问题,具体修改方案是 。
(3)融合蛋白表达成功后,将FLAG-P、FLAG-P△、药物A和UBC按照图乙中的组合方式分成5组。各组样品混匀后分别流经含FLAG抗体的介质,分离出与介质结合的物质并用UBC抗体检测,检测结果如图丙所示。已知FLAG-P和FLAG-P△不能降解UBC,由①②③组结果的差异推测,药物A的作用是 ;由②④组或③⑤组的差异推测,P△中缺失的特定序列的作用是 。
(4)根据以上结果推测,药物A治疗该病的机理是 。
限时集训(十四)B
1.C [解析] ①②③④均为限制酶,可催化磷酸二酯键的断裂,不能催化氢键的断裂,且均形成黏性末端,A错误;①切出的 DNA 片段带有黏性末端,用E.coli DNA连接酶和T4DNA连接酶均能连接起来,B错误;②③切出的黏性末端相同,用DNA连接酶连接后形成的片段的碱基序列为—GGATCT—,因而不能再被②或③识别切割,C正确;构建基因表达载体时,用②③进行双酶切,不能防止目的基因与载体的反向连接,因为这两种限制酶切出的黏性末端是相同的,D错误。
2.D [解析] 可以用Ca2+处理大肠杆菌细胞,使其易于吸收周围环境中的DNA分子(使大肠杆菌处于感受态),有利于重组的基因表达载体导入其中,提高转化效率,A正确;将目的基因导入受体细胞前必须构建基因表达载体,这是基因工程的核心步骤,B正确;花粉管通道法:可以用微量注射器将含目的基因的DNA溶液直接注入子房中,也可以在植物受粉后的一定时间内,剪去柱头,将DNA溶液滴加在花柱切面上,使目的基因借助花粉管通道进入胚囊,C正确;农杆菌转化法总体上有两次拼接过程:发生在Ti质粒与目的基因之间(体外拼接)、携带目的基因的T-DNA与植物细胞的染色体DNA之间(体内拼接),两次导入过程:基因表达载体导入农杆菌、农杆菌T-DNA导入植物细胞,D错误。
3.D [解析] 若线状重组黏粒两端的黏性末端能碱基互补配对,则线状重组黏粒进入受体菌后可能会发生自身环化,A错误;ori是复制原点,不能进行筛选,AmpR序列便于筛选出含目的基因的受体菌,B错误;步骤①表示通过限制酶切割获得具有黏性末端的黏粒和目的基因,一般应使用两种限制酶,防止目的基因与黏粒反向连接,步骤②表示将黏粒和目的基因连接,应使用DNA连接酶,C错误;含目的基因的重组黏粒与λ噬菌体包装蛋白混合后用于构建λ噬菌体,然后用于侵染细菌,将目的基因导入受体菌,D正确。
4.D [解析] 胰蛋白酶和胰凝乳蛋白酶为两种消化酶,蛋白酶抑制剂的抗虫机制是阻断或减弱消化酶作用,导致害虫无法对外来蛋白进行消化作用而发育异常或死亡,A正确;农杆菌能侵染双子叶植物和裸子植物,而对大多数单子叶植物没有侵染能力,因此,利用农杆菌转化法进行转化操作时,不足之处为该方法不适用于单子叶植物,B正确;结合实验结果可看出,NaPⅠ+StPin1A组虫体质量最小,每株棉铃数量最多,故而抗虫效果最佳,C正确;由数据处理图示可以看出,此实验中的自变量有转基因植株的种类和饲喂时间,因变量是虫体质量和每株棉铃数,D错误。
5.(1)NgoMⅣ Ca2+
(2)X 需要 Y 质粒重新环化或质粒间发生自身环化,使得大肠杆菌同时具备基因XR和YR
(3)甲和丙 目的基因反向连接
[解析] (1)由目的基因形成的黏性末端可知,只有NgoM Ⅳ酶切割后的黏性末端,可以与目的基因形成的黏性末端互补配对,因此若要将图B所示的目的基因直接插入YR内形成重组质粒,则质粒需用限制酶NgoM Ⅳ切开,将切开的质粒溶液与目的基因溶液混合后连接,一般先用Ca2+处理大肠杆菌后,使其处于感受态,再对其进行导入操作。(2)质粒中的X抗生素抗性基因XR未被破坏,Y抗生素抗性基因YR由于插入目的基因而被破坏,因此取导入处理过的大肠杆菌接种到含有抗生素X的培养基中培养,为获得纯种,应在固体培养基上培养,培养基需要加入琼脂。Y抗生素抗性基因(YR)由于插入目的基因而被破坏,因此取部分菌体接种到含有抗生素Y的培养基中,若无菌落生长,可初步确定获得的培养物符合要求;若有菌落生长,说明质粒重新环化或质粒间发生自身环化,使得大肠杆菌同时具备基因XR和YR。(3)由图C可知,若要扩增出450 bp片段,且含有目的基因,只能选择引物甲和丙进行PCR。由“扩增出了400 bp片段”,即300 bp和100 bp两部分,推测可能是目的基因发生了反向连接。
6.(1)能与P基因母链的一段碱基序列互补配对、短单链核酸 5'端
(2)P基因编码链的第一个碱基与EcoR Ⅰ识别序列的最后两个碱基编码一个氨基酸,导致mRNA的密码子被错位读取 在引物中的EcoRⅠ识别序列3'端添加1个碱基
(3)增强FLAG-P与UBC的结合 参与P与UBC的结合
(4)药物A通过增强P与UBC结合促进P降解
[解析] (1)引物是一小段能与模板链的一段碱基序列互补配对的短核苷酸单链,设计扩增P基因的引物,需要两种引物分别与两条模板链3'端的碱基序列互补。DNA聚合酶延伸子链时,是将脱氧核苷酸加到引物的3'端,为了不破坏目的基因,该限制酶识别序列应添加在引物的5'端。(2)融合基因转录出的mRNA序列正确,翻译出的融合蛋白中FLAG的氨基酸序列正确,但P基因对应的氨基酸序列与P不同,说明P基因翻译时出错。由图可看出,在转录出的mRNA中,限制酶EcoRⅠ识别序列对应的最后两个碱基与P基因对应的第一个碱基构成一个密码子,导致读码框改变,翻译出的氨基酸序列改变,可通过在EcoRⅠ识别序列3'端增加1个碱基使其碱基数目加上FLAG的碱基数目为3的倍数,这样能保证P基因转录出的mRNA上的读码框不改变,能够正常翻译。(3)①组仅添加UBC,处理后,用UBC抗体检测,不出现杂交带(因为UBC不能与介质结合);②组添加UBC和FLAG-P,用UBC抗体检测,出现杂交带;③组添加UBC、药物A和FLAG-P,用UBC抗体检测,杂交带更加明显,说明药物A的作用是促进UBC与FLAG-P的结合。②④组或③⑤组的差异在于②③组使用FLAG-P,出现杂交带,④⑤组使用FLAG-P△,不出现杂交带,据此推测P△中缺失的特定序列是与UBC结合的关键序列。(4)根据(3)的分析推测,药物A促进UBC与FLAG-P的结合,从而促进蛋白P被蛋白酶识别并降解,达到治疗的目的。
同课章节目录