转录
图片预览










文档简介
(共77张PPT)
第三章 从DNA到RNA——转录
在RNA聚合酶的催化下,以一段DNA链为模板合成RNA,从而将DNA所携带的遗传信息传递给RNA的过程称为转录(Transcription)。
RNA有多种,主要有rRNA,tRNA,mRNA,除此之外还有大量的小RNA。
第一节 转录的特征
一、不对称性
二、单向性
三、连续性
四、有特定的起始和终止位点
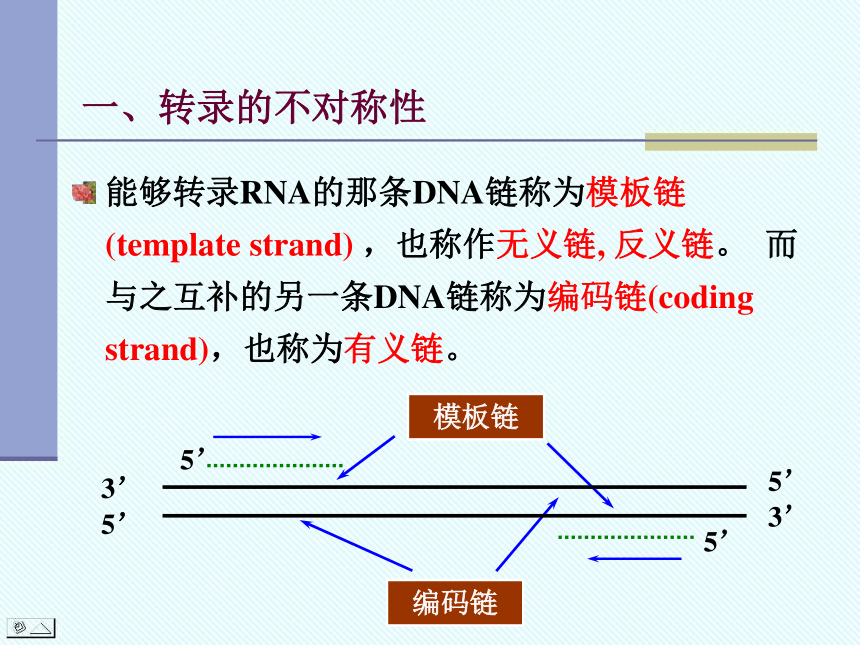
能够转录RNA的那条DNA链称为模板链(template strand) ,也称作无义链, 反义链。 而与之互补的另一条DNA链称为编码链(coding strand),也称为有义链。
一、转录的不对称性
模板链
编码链
5’
5’
3’
3’
5’
5’
5′···GCAGTACATGTC ···3′
3′··· c g t g a t g t a c a g ···5′
编码链
模板链
mRNA
5′···GCAGUACAUGUC ···3′
转录
N······Ala · Val · His · Val ······C
蛋白质
翻译
模板链、编码链与转录及翻译的关系
5
3
3
5
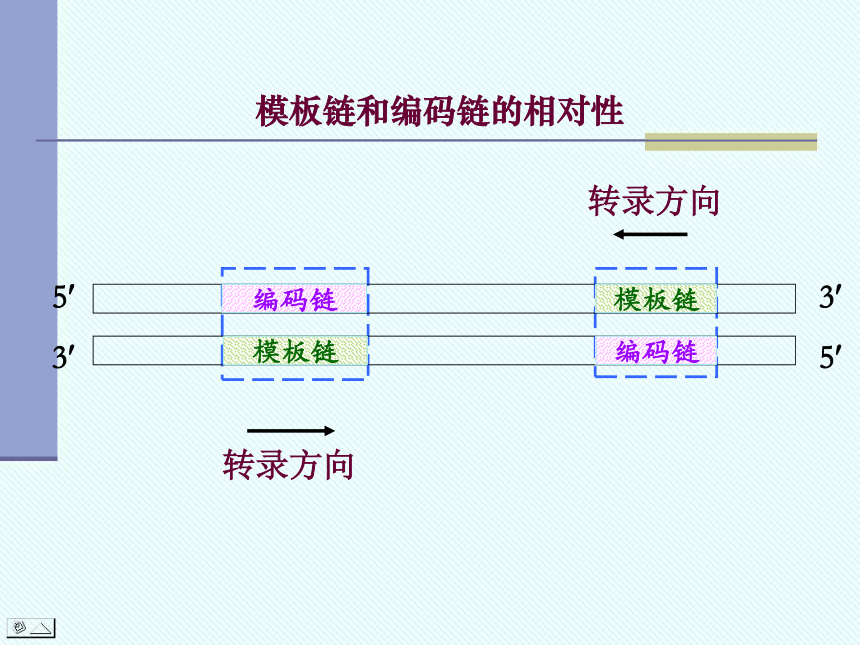
模板链
编码链
编码链
模板链
转录方向
转录方向
模板链和编码链的相对性
RNA转录合成时,在RNA聚合酶的催化下,连续合成一段RNA链,合成过程中没有缺口的填补与连接。
二、转录的连续性
RNA转录时,只能向一个方向进行聚合,所依赖的模板DNA链的方向为3'→5',而RNA链的合成方向为5'→3'。
三、转录的单向性
RNA转录合成时,只以DNA分子中的某一段作为模板。转录事件的发生存在特定的起始位点和特定的终止位点。
特定起始点和特定终止点之间的DNA链构成一个转录单位,通常由转录区和有关的调节顺序构成。
四、有特定的起始和终止位点
第二节 转录机器的主要成分
一、RNA聚合酶
二、转录因子
三、终止因子
是一种以DNA为模板的RNA聚合酶。
该酶在单链DNA模板以及四种三磷酸核苷酸存在的条件下,不需要引物,即可从5'→3'聚合RNA。
一、RNA聚合酶(RNA pol)
原核生物中的RNA聚合酶全酶由五个亚基构成,即 2 ' 。
亚基与转录起始点的识别有关,在转录合成开始后被释放;余下的部分( 2 ‘)被称为核心酶,与RNA链的聚合有关。
(一)原核生物的RNA聚合酶
核心酶 (core enzyme)
全酶 (holoenzyme)
原核生物RNA聚合酶的全酶和核心酶
大肠杆菌有不同的 因子,分别识别不同的启动子。
枯草杆菌 因子的更替:枯草杆菌噬菌体SPO1能合成替代宿主菌 因子的蛋白质,使RNA聚合酶识别病毒启动子。
真核生物中的RNA聚合酶可按其对 -鹅膏蕈碱的敏感性而分为三种,它们均由10~12个大小不同的亚基所组成,结构非常复杂,其功能也不同。
(二)真核生物的RNA聚合酶
在真核生物中,转录的起始过程较为复杂。现已发现数百种蛋白因子与RNA转录合成有关,这些蛋白因子被统称为反式作用因子(trans-acting factor)。
在反式作用因子中,直接或间接参与转录起始复合体的形成的蛋白因子被称为转录因子(transcriptional factor, TF)。
二、转录因子
真核生物RNA聚合酶Ⅱ转录因子及其功能
原核生物中的终止因子ρ蛋白是一种六聚体的蛋白质,亚基的分子量为50kd,有NTP酶活性。
该蛋白因子能识别转录终止信号,导致RNA的释放与RNA聚合酶的解离。
三、终止因子
第三节 启动子与转录起始
一、原核启动子与转录起始
二、真核启动子与转录起始
启动子(Promoter)指能被RNA聚合酶识别、结合并启动基因转录的一段DNA序列。
转录起始:(1)RNA聚合酶结合在转录模板的起始区域(启动子)。(2) DNA双链解开,使其中的一条链作为转录的模板。
与新生RNA链第一个核苷酸对应DNA链上的碱基称为转录起点,起点为+1,起点前面称为上游,依次标记为-1,-2……。
一、原核启动子与转录起始
大肠杆菌RNA聚合酶与启动子相互作用包括启动子区的识别、酶与启动子的结合及 因子的解离。
原核启动子区的发现——Pribnow实验
DNA酶I 水解
分离
DNA片段
开始转录
(Pribnow box)
T A T A A T Pu A T A T T A Py
-10 区
1
-30
-50
10
-10
-40
-20
5
3
3
5
T T G A C A
A A C T G T
-35 区
RNA-pol辨认位点
(recognition site)
5
5
RNA聚合酶保护区
结构基因
3
3
原核启动子区的发现——Pribnow实验
原核生物转录起始区的一致性序列
被RNA聚合酶辨认的区段就是位于转录起始点-35区的TTGACA序列。
酶与该区结合后,即滑动至-10区的TATAAT序列(Pribnow盒) ,并启动转录。
RNA聚合酶全酶在转录起始区的结合
⑵ DNA局部双链解开。
⑴ RNA聚合酶全酶( 2 )与模板结合。
⑶ 在RNA聚合酶作用下发生第一次聚合反应,形成转录起始复合物。
RNApol ( 2 ) - DNA - pppGpN- OH 3
转录起始复合物:三元复合物
5 -pppG -OH + NTP 5 -pppGpN - OH 3 + ppi
原核转录起始过程
二、真核启动子与转录起始
真核生物RNA聚合酶II的转录起始点上游-25bp区也存在一段富含TA的顺序,被称为TATA盒(TATA Box),通常认为是启动子的核心序列。
除此之外,在真核生物中还可见到其他带共性的上游序列,如CAAT盒及GC盒等。
在远离受控基因的调控序列称为增强子(Enhancer)。
真核生物启动子保守序列
真核生物mRNA转录起始时,首先由TFⅡD的TBP亚基识别并结合TATA盒,然后在其他转录因子的配合下,与RNA聚合酶Ⅱ组装形成转录起始前复合物(pre-initiation complex, PIC)。
RNA聚合酶Ⅱ催化第一个磷酸二酯键形成。
RNA聚合酶Ⅱ的羧基末端结构域(CTD)被磷酸化修饰,大部分转录因子脱离,聚合酶向下游移动延伸RNA链。
真核mRNA转录起始过程
真核生物RNA聚合酶Ⅱ转录因子及其功能
RNA聚合酶II 大亚基中有 C 末端结构域 (carboxy terminal domain CTD)
CTD中含一保守氨基酸序列的多个重复
Tyr-Ser-Pro-Thr-Ser-Pro-Ser
C 端重复七肽
其中Ser可以被磷酸化。
POL-Ⅱ
TFⅡF
ⅡA
ⅡB
POL-Ⅱ
TFⅡF
ⅡH
ⅡE
TBP
TAF
TFⅡD-ⅡA-ⅡB-DNA复合物
TATA
ⅡA
ⅡB
TBP
TAF
TATA
ⅡH
ⅡE
CTD-
P
PIC组装完成,TFⅡH使CTD磷酸化
部分RNA聚合酶III识别的启动子在转录起始位点下游,即编码区内部,称为内部启动子或下游启动子。
5SrRNA基因与tRNA基因为内部启动子
内部启动子
tRNA基因内部启动子
第四节 转录延伸与终止
一、转录延伸
三、终止机制
四、抗终止
二、终止与终止子
一、转录延伸
亚基脱落,RNA–pol聚合酶核心酶变构,与模板结合松弛,沿着DNA模板前移;
在核心酶作用下,NTP不断聚合,RNA链不断延长。
原核转录延伸
(NMP) n + NTP (NMP) n+1 + PPi
原核生物转录过程中的羽毛状现象
真核生物转录延长过程与原核生物类似,但由于存在核小体的高级结构,故在转录延长过程中可观察到核小体移位和解聚现象。
真核转录延伸
RNA聚合酶停止把底物逐个加到RNA 3’-OH 释放出完成的RNA链 酶从模板上解离
终止子(Terminator)是模板DNA上的终止信号,由它转录产生的RNA决定了转录的终止。
原核终止子的类别
不依赖ρ因子:可以独立完成终止作用
依赖ρ因子:终止作用需要ρ因子的辅助。
二、终止与终止子
上游有1个富含GC对的二重对称区(反向互补序列),转录后的RNA可形成发夹结构。
终止位点前有一段4-8个A组成的序列,形成寡聚U的转录产物。
不依赖ρ因子的终止子
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
RNA
5 TTGCAGCCTGACAAATCAGGCTGATGGCTGGTGACTTTTTAGTCACCAGCCTTTTT... 3
DNA
UUUUU...…
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
茎环(stem-loop)/发夹(hairpin)结构
结构与不依赖ρ因子的终止子相似,但是序列存在差异,表现为对称区GC含量较少,后面无连续的A。
依赖ρ因子的终止子
无连续 U
G/C含量较少
不依赖ρ因子的终止:
RNA转录产物形成发夹结构,引起RNA聚合酶暂停,寡聚U提供解离信号,导致RNA转录的终止,RNA-DNA解离,RNA聚合酶释放。
三、终止机制
依赖ρ因子的终止
ρ因子的作用
a、ρ因子与RNA结合
b、ρ因子沿RNA从5’→3’移动(NTP水解供能)
c、ρ因子与RNA聚合酶相互作用造成转录的终止
因子:六聚体蛋白、水解各种核甘三磷酸促使新生RNA链从三元转录复合物中解离出来,从而终止转录。
ρ因子结合并追赶RNApol
RNApol暂停, ρ因子赶上RNApol与RNApol相互作用
三元复合体解离
真核生物mRNA的转录终止
5 ------AAUAAA-
5 ------AAUAAA--
核酸酶
-GUGUGUG
RNA-pol
AATAAA GTGTGTG
转录终止的修饰点
5
5
3
3
3 加尾
真核生物转录终止与poly A尾巴结构的添加密切相关。在poly A修饰位点的附近存在一组共同序列AATAAA和GTGTGT……,为转录终止的识别修饰位点。
转录过程中遇到终止信号,仍然继续转录,称为抗终止现象。
抗终止的两种机制
(1)破坏终止位点RNA的发夹结构
(2)依赖于蛋白质因子的转录抗终止
四、抗终止
核糖体破坏发夹结构的形成
ρ因子
ρ因子
核糖体
核糖体滞留
核糖体滞留在发夹区破坏发夹结构的形成
第五节 RNA的转录后加工
一、戴帽与加尾
二、内含子的切除
三、RNA编辑
四、核苷酸修饰
1.戴帽(adding cap):
真核细胞mRNA转录后会在5’-端加上m7GTP结构。戴帽的过程发生在细胞核内。
帽子结构的意义 :
(1) 对翻译起识别作用------为核糖体识别RNA提供信号
(2) 增加mRNA 的稳定性,使5’端免遭外切核酸酶的攻击
一、戴帽与加尾
Pi
5 ppGp…
磷酸酶
5 pppGp…
5 GpppGp…
pppG
ppi
鸟苷酸转移酶
5 m7GpppGp…
甲基转移酶
SAM
帽子结构的生成
2.加尾(adding tail):
真核细胞mRNA转录终止后,首先由核酸外切酶切去3'端一些过剩的核苷酸,然后再加入polyA。
加尾的意义:
(1)polyA结构与mRNA的半衰期有关。
(2)可能与核质转运有关
加尾的位点与识别序列
5 ------AAUAAA-
5 ------AAUAAA--
核酸酶
-GUGUGUG
RNA-pol
AATAAA GTGTGTG
转录终止的修饰点
5
5
3
3
3 加尾
鸡卵清蛋白成熟mRNA与DNA杂交电镜图
mRNA
DNA
二、RNA的剪接
1. 断裂基因(splite gene)
B
A
C
编码区 A、B、C
非编码区
真核生物结构基因,由若干个编码区和非编码区互相间隔,去除非编码区再连接后,可翻译出由连续氨基酸组成的完整蛋白质,这些基因称为断裂基因。
外显子(exon)和内含子(intron)
外显子
结构基因中多肽链的编码序列
内含子
结构基因中的非编码序列,在mRNA成熟过程中被切除。
RNA剪接
去除原始转录产物——hnRNA中的内含子,将外显子拼接成成熟mRNA的过程。
鸡卵清蛋
白基因
hnRNA
首、尾修饰
hnRNA剪接
成熟的mRNA
鸡卵清蛋白基因及其转录、转录后修饰
RNA剪接方式的多样性
RNA剪接方式主要可分为三类:
机制一:由拼接装置完成(核mRNA内含子)
可供识别的特异序列
拼接装置由多种蛋白质和核蛋白组成
机制二:自我拼接(两类内含子Ⅰ 、Ⅱ )
形成特定的二级结构
RNA具有催化拼接的能力
机制三:需要蛋白质酶参与的拼接(tRNA内含子)
Breathnach-Chambon rule (GU-AG规则)
● 5’---exon--- GU--------intron--------AG ------exon----3’
供点 100% 94% 受点
● 7Nt 分支位点 3’拼接点的上游
py X py U pu A py
机制一——拼接装置
序列特征:
多数核mRNA前体中内含子5’边界序列是GU,3’边界序列是AG,这一序列模式称为GU-AG规则
snRNA为核内小RNA,富含尿嘧啶,故以U作分类和命名,如U1、U2等。snRNA与核内蛋白质组装形成小核蛋白体(snRNP),参与hnRNA的剪接加工。
snRNP与hnRNA结合成为剪接体。
剪接体结构
机制二——核酶
UpA
GpU
外显子1
内含子
外显子2
pG-OH
(ppG-OH, pppG-OH)
U-OH
GpU
pGpA
第一次转酯反应
第二次转酯反应
G-OH
UpU
pGpA
四膜虫rRNA内含子的二级结构
四膜虫rRNA的剪接采用自身剪接方式
5 -端核苷酸序列
四膜虫前rRNA的自身剪接过程
tRNA前体的转录合成
机制三——核酸内切酶
三、RNA的编辑(RNA editing)
RNA编辑(editing)是指转录后的RNA在编码区发生碱基的突变、加入或丢失等现象。
RNA 编辑现象的发现:1986 R.Benne在研究锥虫线粒体mRNA转录加工时发现mRNA的多个编码位置上加入或丢失尿苷酸,1990年在高等动物和病毒中也发现了编辑现象。
T
U
A
U
A
U
G
UUUU
G
UU
G
UUU
A
UU
A
U
G
U
GA
UU
A
U
GG
UUUU
G
UUUUUU
A
UU
GGUA
UUUUUU
AUA
UUU
A
UUU
AA
UUU
G
UU
GA
U
AAA
U
ACA
UUUU
AUUUG
UU
UG
UU
AA
UUUUUUU
G
UUUU
G
U
GUUUUUGG
UU
TT
TTTT
U
AGG
UUUUUUU
G
UU
GUUG
UU
G
UUUU
G
U
A
UU
A
U
GA
UU
GAG
UUU
G
UU
G
UUU
GG
UUUUUU
G
UUUU
TT
TTTT
UU
G
U
GAAACCAG
UU
AUGAGAGUUUGCA
UU
G
UU
A
UUU
A
UU
ACA
UU
AAG
UU
GGUG
UUUUU
GG
UU
C
图
13-44
锥虫
(
T.brucei
)
cox
Ⅱ
基因的部分
RNA
顺序。很多
U(
红色
)
在
DNA
中未编码,而另一些在
DNA
中编码的
T(
紫色
)
在
mRNA
中被删除了。
(
参考
B.Lewin:
《
GENES
》
Ⅵ
,1997
,
Fig31.16)
锥虫 coxII 基因的编辑
RNA编辑的分子机制
通过gRNA(guide RNA)的指导作用。gRNA可和被辑的前体RNA分子的编辑区序列互补结合。在互补区中gRNA的“A”的相应位置留下了空缺,通过转酯反应,插入“U”。
RNA编辑与RNA剪接异同
相同点:都是转录后的加工形式,增加了遗传信息,都通过转酯反应完成。
不同点:RNA剪接的方式由自身序列决定,RNA编辑的方式由gRNA决定
RNA编辑作用说明,基因的编码序列经过转录后加工,是可有多用途分化的,因此也称为分化加工(differential RNA processing),RNA编辑增加了遗传信息。
四、化学修饰
有些RNA,特别是rRNA与tRNA,有特异的化学修饰。
主要修饰类型:
甲基化
去氨基化(鸟嘌呤 次黄嘌呤)
硫代
核苷酸的替代
二价键的饱和化(二氢尿嘧啶)
同分异构化(尿嘧啶 假尿嘧啶)
核仁RNA(snoRNA)参与RNA的化学修饰,确定修饰位点。
tRNA的碱基修饰
(2)还原反应
如:U DHU
(3)核苷内的转位反应
如:U ψ
(4)脱氨反应
如:A I
如:A Am
(1)甲基化
(1)
(1)
(3)
(2)
(4)
转录的基本特征
RNA聚合酶的种类与功能
原核启动子与真核启动子的特点
原核转录起始与真核转录起始机制
原核终止子的类型、特点与终止机制
戴帽与加尾过程及其生理意义
三种RNA剪接机制
RNA编辑分子机制及生理意义
总结:原核与真核细胞转录过程的区别
本章要点
第三章 从DNA到RNA——转录
在RNA聚合酶的催化下,以一段DNA链为模板合成RNA,从而将DNA所携带的遗传信息传递给RNA的过程称为转录(Transcription)。
RNA有多种,主要有rRNA,tRNA,mRNA,除此之外还有大量的小RNA。
第一节 转录的特征
一、不对称性
二、单向性
三、连续性
四、有特定的起始和终止位点
能够转录RNA的那条DNA链称为模板链(template strand) ,也称作无义链, 反义链。 而与之互补的另一条DNA链称为编码链(coding strand),也称为有义链。
一、转录的不对称性
模板链
编码链
5’
5’
3’
3’
5’
5’
5′···GCAGTACATGTC ···3′
3′··· c g t g a t g t a c a g ···5′
编码链
模板链
mRNA
5′···GCAGUACAUGUC ···3′
转录
N······Ala · Val · His · Val ······C
蛋白质
翻译
模板链、编码链与转录及翻译的关系
5
3
3
5
模板链
编码链
编码链
模板链
转录方向
转录方向
模板链和编码链的相对性
RNA转录合成时,在RNA聚合酶的催化下,连续合成一段RNA链,合成过程中没有缺口的填补与连接。
二、转录的连续性
RNA转录时,只能向一个方向进行聚合,所依赖的模板DNA链的方向为3'→5',而RNA链的合成方向为5'→3'。
三、转录的单向性
RNA转录合成时,只以DNA分子中的某一段作为模板。转录事件的发生存在特定的起始位点和特定的终止位点。
特定起始点和特定终止点之间的DNA链构成一个转录单位,通常由转录区和有关的调节顺序构成。
四、有特定的起始和终止位点
第二节 转录机器的主要成分
一、RNA聚合酶
二、转录因子
三、终止因子
是一种以DNA为模板的RNA聚合酶。
该酶在单链DNA模板以及四种三磷酸核苷酸存在的条件下,不需要引物,即可从5'→3'聚合RNA。
一、RNA聚合酶(RNA pol)
原核生物中的RNA聚合酶全酶由五个亚基构成,即 2 ' 。
亚基与转录起始点的识别有关,在转录合成开始后被释放;余下的部分( 2 ‘)被称为核心酶,与RNA链的聚合有关。
(一)原核生物的RNA聚合酶
核心酶 (core enzyme)
全酶 (holoenzyme)
原核生物RNA聚合酶的全酶和核心酶
大肠杆菌有不同的 因子,分别识别不同的启动子。
枯草杆菌 因子的更替:枯草杆菌噬菌体SPO1能合成替代宿主菌 因子的蛋白质,使RNA聚合酶识别病毒启动子。
真核生物中的RNA聚合酶可按其对 -鹅膏蕈碱的敏感性而分为三种,它们均由10~12个大小不同的亚基所组成,结构非常复杂,其功能也不同。
(二)真核生物的RNA聚合酶
在真核生物中,转录的起始过程较为复杂。现已发现数百种蛋白因子与RNA转录合成有关,这些蛋白因子被统称为反式作用因子(trans-acting factor)。
在反式作用因子中,直接或间接参与转录起始复合体的形成的蛋白因子被称为转录因子(transcriptional factor, TF)。
二、转录因子
真核生物RNA聚合酶Ⅱ转录因子及其功能
原核生物中的终止因子ρ蛋白是一种六聚体的蛋白质,亚基的分子量为50kd,有NTP酶活性。
该蛋白因子能识别转录终止信号,导致RNA的释放与RNA聚合酶的解离。
三、终止因子
第三节 启动子与转录起始
一、原核启动子与转录起始
二、真核启动子与转录起始
启动子(Promoter)指能被RNA聚合酶识别、结合并启动基因转录的一段DNA序列。
转录起始:(1)RNA聚合酶结合在转录模板的起始区域(启动子)。(2) DNA双链解开,使其中的一条链作为转录的模板。
与新生RNA链第一个核苷酸对应DNA链上的碱基称为转录起点,起点为+1,起点前面称为上游,依次标记为-1,-2……。
一、原核启动子与转录起始
大肠杆菌RNA聚合酶与启动子相互作用包括启动子区的识别、酶与启动子的结合及 因子的解离。
原核启动子区的发现——Pribnow实验
DNA酶I 水解
分离
DNA片段
开始转录
(Pribnow box)
T A T A A T Pu A T A T T A Py
-10 区
1
-30
-50
10
-10
-40
-20
5
3
3
5
T T G A C A
A A C T G T
-35 区
RNA-pol辨认位点
(recognition site)
5
5
RNA聚合酶保护区
结构基因
3
3
原核启动子区的发现——Pribnow实验
原核生物转录起始区的一致性序列
被RNA聚合酶辨认的区段就是位于转录起始点-35区的TTGACA序列。
酶与该区结合后,即滑动至-10区的TATAAT序列(Pribnow盒) ,并启动转录。
RNA聚合酶全酶在转录起始区的结合
⑵ DNA局部双链解开。
⑴ RNA聚合酶全酶( 2 )与模板结合。
⑶ 在RNA聚合酶作用下发生第一次聚合反应,形成转录起始复合物。
RNApol ( 2 ) - DNA - pppGpN- OH 3
转录起始复合物:三元复合物
5 -pppG -OH + NTP 5 -pppGpN - OH 3 + ppi
原核转录起始过程
二、真核启动子与转录起始
真核生物RNA聚合酶II的转录起始点上游-25bp区也存在一段富含TA的顺序,被称为TATA盒(TATA Box),通常认为是启动子的核心序列。
除此之外,在真核生物中还可见到其他带共性的上游序列,如CAAT盒及GC盒等。
在远离受控基因的调控序列称为增强子(Enhancer)。
真核生物启动子保守序列
真核生物mRNA转录起始时,首先由TFⅡD的TBP亚基识别并结合TATA盒,然后在其他转录因子的配合下,与RNA聚合酶Ⅱ组装形成转录起始前复合物(pre-initiation complex, PIC)。
RNA聚合酶Ⅱ催化第一个磷酸二酯键形成。
RNA聚合酶Ⅱ的羧基末端结构域(CTD)被磷酸化修饰,大部分转录因子脱离,聚合酶向下游移动延伸RNA链。
真核mRNA转录起始过程
真核生物RNA聚合酶Ⅱ转录因子及其功能
RNA聚合酶II 大亚基中有 C 末端结构域 (carboxy terminal domain CTD)
CTD中含一保守氨基酸序列的多个重复
Tyr-Ser-Pro-Thr-Ser-Pro-Ser
C 端重复七肽
其中Ser可以被磷酸化。
POL-Ⅱ
TFⅡF
ⅡA
ⅡB
POL-Ⅱ
TFⅡF
ⅡH
ⅡE
TBP
TAF
TFⅡD-ⅡA-ⅡB-DNA复合物
TATA
ⅡA
ⅡB
TBP
TAF
TATA
ⅡH
ⅡE
CTD-
P
PIC组装完成,TFⅡH使CTD磷酸化
部分RNA聚合酶III识别的启动子在转录起始位点下游,即编码区内部,称为内部启动子或下游启动子。
5SrRNA基因与tRNA基因为内部启动子
内部启动子
tRNA基因内部启动子
第四节 转录延伸与终止
一、转录延伸
三、终止机制
四、抗终止
二、终止与终止子
一、转录延伸
亚基脱落,RNA–pol聚合酶核心酶变构,与模板结合松弛,沿着DNA模板前移;
在核心酶作用下,NTP不断聚合,RNA链不断延长。
原核转录延伸
(NMP) n + NTP (NMP) n+1 + PPi
原核生物转录过程中的羽毛状现象
真核生物转录延长过程与原核生物类似,但由于存在核小体的高级结构,故在转录延长过程中可观察到核小体移位和解聚现象。
真核转录延伸
RNA聚合酶停止把底物逐个加到RNA 3’-OH 释放出完成的RNA链 酶从模板上解离
终止子(Terminator)是模板DNA上的终止信号,由它转录产生的RNA决定了转录的终止。
原核终止子的类别
不依赖ρ因子:可以独立完成终止作用
依赖ρ因子:终止作用需要ρ因子的辅助。
二、终止与终止子
上游有1个富含GC对的二重对称区(反向互补序列),转录后的RNA可形成发夹结构。
终止位点前有一段4-8个A组成的序列,形成寡聚U的转录产物。
不依赖ρ因子的终止子
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
RNA
5 TTGCAGCCTGACAAATCAGGCTGATGGCTGGTGACTTTTTAGTCACCAGCCTTTTT... 3
DNA
UUUUU...…
5`UUGCAGCCUGACAAAUCAGGCUGAUGGCUGGUGACUUUUUAGUCACCAGCCUUUUU... 3`
茎环(stem-loop)/发夹(hairpin)结构
结构与不依赖ρ因子的终止子相似,但是序列存在差异,表现为对称区GC含量较少,后面无连续的A。
依赖ρ因子的终止子
无连续 U
G/C含量较少
不依赖ρ因子的终止:
RNA转录产物形成发夹结构,引起RNA聚合酶暂停,寡聚U提供解离信号,导致RNA转录的终止,RNA-DNA解离,RNA聚合酶释放。
三、终止机制
依赖ρ因子的终止
ρ因子的作用
a、ρ因子与RNA结合
b、ρ因子沿RNA从5’→3’移动(NTP水解供能)
c、ρ因子与RNA聚合酶相互作用造成转录的终止
因子:六聚体蛋白、水解各种核甘三磷酸促使新生RNA链从三元转录复合物中解离出来,从而终止转录。
ρ因子结合并追赶RNApol
RNApol暂停, ρ因子赶上RNApol与RNApol相互作用
三元复合体解离
真核生物mRNA的转录终止
5 ------AAUAAA-
5 ------AAUAAA--
核酸酶
-GUGUGUG
RNA-pol
AATAAA GTGTGTG
转录终止的修饰点
5
5
3
3
3 加尾
真核生物转录终止与poly A尾巴结构的添加密切相关。在poly A修饰位点的附近存在一组共同序列AATAAA和GTGTGT……,为转录终止的识别修饰位点。
转录过程中遇到终止信号,仍然继续转录,称为抗终止现象。
抗终止的两种机制
(1)破坏终止位点RNA的发夹结构
(2)依赖于蛋白质因子的转录抗终止
四、抗终止
核糖体破坏发夹结构的形成
ρ因子
ρ因子
核糖体
核糖体滞留
核糖体滞留在发夹区破坏发夹结构的形成
第五节 RNA的转录后加工
一、戴帽与加尾
二、内含子的切除
三、RNA编辑
四、核苷酸修饰
1.戴帽(adding cap):
真核细胞mRNA转录后会在5’-端加上m7GTP结构。戴帽的过程发生在细胞核内。
帽子结构的意义 :
(1) 对翻译起识别作用------为核糖体识别RNA提供信号
(2) 增加mRNA 的稳定性,使5’端免遭外切核酸酶的攻击
一、戴帽与加尾
Pi
5 ppGp…
磷酸酶
5 pppGp…
5 GpppGp…
pppG
ppi
鸟苷酸转移酶
5 m7GpppGp…
甲基转移酶
SAM
帽子结构的生成
2.加尾(adding tail):
真核细胞mRNA转录终止后,首先由核酸外切酶切去3'端一些过剩的核苷酸,然后再加入polyA。
加尾的意义:
(1)polyA结构与mRNA的半衰期有关。
(2)可能与核质转运有关
加尾的位点与识别序列
5 ------AAUAAA-
5 ------AAUAAA--
核酸酶
-GUGUGUG
RNA-pol
AATAAA GTGTGTG
转录终止的修饰点
5
5
3
3
3 加尾
鸡卵清蛋白成熟mRNA与DNA杂交电镜图
mRNA
DNA
二、RNA的剪接
1. 断裂基因(splite gene)
B
A
C
编码区 A、B、C
非编码区
真核生物结构基因,由若干个编码区和非编码区互相间隔,去除非编码区再连接后,可翻译出由连续氨基酸组成的完整蛋白质,这些基因称为断裂基因。
外显子(exon)和内含子(intron)
外显子
结构基因中多肽链的编码序列
内含子
结构基因中的非编码序列,在mRNA成熟过程中被切除。
RNA剪接
去除原始转录产物——hnRNA中的内含子,将外显子拼接成成熟mRNA的过程。
鸡卵清蛋
白基因
hnRNA
首、尾修饰
hnRNA剪接
成熟的mRNA
鸡卵清蛋白基因及其转录、转录后修饰
RNA剪接方式的多样性
RNA剪接方式主要可分为三类:
机制一:由拼接装置完成(核mRNA内含子)
可供识别的特异序列
拼接装置由多种蛋白质和核蛋白组成
机制二:自我拼接(两类内含子Ⅰ 、Ⅱ )
形成特定的二级结构
RNA具有催化拼接的能力
机制三:需要蛋白质酶参与的拼接(tRNA内含子)
Breathnach-Chambon rule (GU-AG规则)
● 5’---exon--- GU--------intron--------AG ------exon----3’
供点 100% 94% 受点
● 7Nt 分支位点 3’拼接点的上游
py X py U pu A py
机制一——拼接装置
序列特征:
多数核mRNA前体中内含子5’边界序列是GU,3’边界序列是AG,这一序列模式称为GU-AG规则
snRNA为核内小RNA,富含尿嘧啶,故以U作分类和命名,如U1、U2等。snRNA与核内蛋白质组装形成小核蛋白体(snRNP),参与hnRNA的剪接加工。
snRNP与hnRNA结合成为剪接体。
剪接体结构
机制二——核酶
UpA
GpU
外显子1
内含子
外显子2
pG-OH
(ppG-OH, pppG-OH)
U-OH
GpU
pGpA
第一次转酯反应
第二次转酯反应
G-OH
UpU
pGpA
四膜虫rRNA内含子的二级结构
四膜虫rRNA的剪接采用自身剪接方式
5 -端核苷酸序列
四膜虫前rRNA的自身剪接过程
tRNA前体的转录合成
机制三——核酸内切酶
三、RNA的编辑(RNA editing)
RNA编辑(editing)是指转录后的RNA在编码区发生碱基的突变、加入或丢失等现象。
RNA 编辑现象的发现:1986 R.Benne在研究锥虫线粒体mRNA转录加工时发现mRNA的多个编码位置上加入或丢失尿苷酸,1990年在高等动物和病毒中也发现了编辑现象。
T
U
A
U
A
U
G
UUUU
G
UU
G
UUU
A
UU
A
U
G
U
GA
UU
A
U
GG
UUUU
G
UUUUUU
A
UU
GGUA
UUUUUU
AUA
UUU
A
UUU
AA
UUU
G
UU
GA
U
AAA
U
ACA
UUUU
AUUUG
UU
UG
UU
AA
UUUUUUU
G
UUUU
G
U
GUUUUUGG
UU
TT
TTTT
U
AGG
UUUUUUU
G
UU
GUUG
UU
G
UUUU
G
U
A
UU
A
U
GA
UU
GAG
UUU
G
UU
G
UUU
GG
UUUUUU
G
UUUU
TT
TTTT
UU
G
U
GAAACCAG
UU
AUGAGAGUUUGCA
UU
G
UU
A
UUU
A
UU
ACA
UU
AAG
UU
GGUG
UUUUU
GG
UU
C
图
13-44
锥虫
(
T.brucei
)
cox
Ⅱ
基因的部分
RNA
顺序。很多
U(
红色
)
在
DNA
中未编码,而另一些在
DNA
中编码的
T(
紫色
)
在
mRNA
中被删除了。
(
参考
B.Lewin:
《
GENES
》
Ⅵ
,1997
,
Fig31.16)
锥虫 coxII 基因的编辑
RNA编辑的分子机制
通过gRNA(guide RNA)的指导作用。gRNA可和被辑的前体RNA分子的编辑区序列互补结合。在互补区中gRNA的“A”的相应位置留下了空缺,通过转酯反应,插入“U”。
RNA编辑与RNA剪接异同
相同点:都是转录后的加工形式,增加了遗传信息,都通过转酯反应完成。
不同点:RNA剪接的方式由自身序列决定,RNA编辑的方式由gRNA决定
RNA编辑作用说明,基因的编码序列经过转录后加工,是可有多用途分化的,因此也称为分化加工(differential RNA processing),RNA编辑增加了遗传信息。
四、化学修饰
有些RNA,特别是rRNA与tRNA,有特异的化学修饰。
主要修饰类型:
甲基化
去氨基化(鸟嘌呤 次黄嘌呤)
硫代
核苷酸的替代
二价键的饱和化(二氢尿嘧啶)
同分异构化(尿嘧啶 假尿嘧啶)
核仁RNA(snoRNA)参与RNA的化学修饰,确定修饰位点。
tRNA的碱基修饰
(2)还原反应
如:U DHU
(3)核苷内的转位反应
如:U ψ
(4)脱氨反应
如:A I
如:A Am
(1)甲基化
(1)
(1)
(3)
(2)
(4)
转录的基本特征
RNA聚合酶的种类与功能
原核启动子与真核启动子的特点
原核转录起始与真核转录起始机制
原核终止子的类型、特点与终止机制
戴帽与加尾过程及其生理意义
三种RNA剪接机制
RNA编辑分子机制及生理意义
总结:原核与真核细胞转录过程的区别
本章要点
同课章节目录
- 前言
- 第一章 孟德尔定律
- 第一节 分离定律
- 第二节 自由组合定律
- 第二章 染色体与遗传
- 第一节 减数分裂中的染色体行为
- 第二节 遗传的染色体学说
- 第三节 性染色体与伴性遗传
- 第三章 遗传的分子基础
- 第一节 核酸是遗传物质的证据
- 第二节 DNA的分子结构和特点
- 第三节 遗传信息的传递
- 第四节 遗传信息的表达—-RNA和蛋白质的合成
- 第四章 生物的变异
- 第一节 生物变异的来源
- 第二节 生物变异在生产上的应用
- 第五章 生物的进化
- 第一节 生物的多样性、统一性和进化
- 第二节 进化性变化是怎样发生的
- 第三节 探索生物进化的历史
- 第六章 遗传与人类健康
- 第一节 人类遗传病的主要类型
- 第二节 遗传咨询与优生
- 第三节 基因治疗和人类基因组计划
- 第四节 遗传病与人类未来